【论著】| 循环肿瘤细胞FCGBP和BIGH3作为结直肠癌潜在生物标志物的可行性研究
2024-10-17 中国癌症杂志 中国癌症杂志
通过结直肠癌组织验证表达以及肿瘤功能实验证实BIGH3与结直肠癌相关,可能是潜在的治疗靶点。
[摘要] 背景与目的:结直肠癌(colorectal cancer,CRC)是全球常见的恶性肿瘤之一,且晚期患者的预后较差,寻找新的CRC潜在生物标志物的需求日益增加。循环肿瘤细胞(circulating tumor cell,CTC)从原发肿瘤脱落并进入循环系统,可在血液中被检测到,被认为是CRC的重要生物标志物。本研究旨在探讨CRC中的FCGBP和BIGH3是否可作为结直肠癌的潜在生物标志物。方法:本研究从基因表达综合数据库(Gene Expression Omnibus,GEO)获取了3个有CTC的CRC数据集,分别为GSE74369、GSE117606和GSE164191。通过生物信息学分析在CTC和正常样本之间筛选出差异表达基因。其中一个包含临床信息的数据集用于WGCNA分析,并鉴定了两个关键基因模块,共包含1 148个基因。然后对这些模块中的基因进行了功能富集分析。使用Venn图、PPI调控网络构建分析和筛选候选基因。最后,使用癌症基因组图谱(The Cancer Genome Atlas,TCGA)和基因型-组织表达数据库(Genotype-Tissue Expression,GTEx)数据进行生存分析,并鉴定出与CRC相关的关键基因。在结直肠癌组织中通过实时荧光定量聚合酶链反应(real-time fluorescence quantitative polymerase chain reaction,RTFQ-PCR)验证BIGH3基因表达,并通过克隆形成、划痕实验以及transwell实验等功能实验在HCT116和SW620细胞系中验证BIGH3基因与结直肠癌的关系。结果:通过GEO中CRC数据集分析,共筛选出了2 214个差异基因,通过WGCNA分析和PPI网络构建鉴定出4个与CRC相关的CTC基因。通过GEPIA数据库进行生存分析发现, FCGBP及BIGH3与总生存期及无疾病生存期具有相关性。进一步实验表明,BIGH3基因在30对配对的结直肠癌样本中呈高表达,在HCT116和SW620细胞系中敲低BIGH3的表达能够减慢细胞增殖和迁移的速度,并降低侵袭性,而上调BIGH3的表达则会增加其侵袭性或提高迁移率。结论:本研究结果表明,FCGBP及BIGH3与TNM分期呈正相关,这暗示它们在CRC发展中的重要作用,具有较好的预后价值,其可以作为潜在的CRC生物标志物,并可能作为潜在的治疗靶点。同时,我们的实验数据也揭示了BIGH3在结直肠癌中的重要角色,它可能影响结直肠癌的生物学行为。
[关键词] 结直肠癌;循环肿瘤细胞;生物标志物
[Abstract] Background and purpose: Colorectal cancer (CRC) is globally recognized as one of the most prevalent malignant tumors. Advanced CRC is marked by a relatively poor prognosis for patients, signifying an urgent need to identify novel potential biomarkers for CRC. A particular focus has been given to circulating tumor cells (CTC), which are cells that have detached from the primary tumor mass and subsequently entered the circulatory system. These cells can be detected within the blood and are currently considered significant potential biomarkers for CRC. This study aimed to investigate if FCGBP and BIGH3 in CRC could be potential markers for colorectal cancer. Methods: This study obtained 3 CRC datasets (GSE74369, GSE117606, and GSE164191) with CTC from the GEO database. Bioinformatics analysis was conducted to screen differentially expressed genes between CTC and normal samples. One dataset that includes clinical information was used for WGCNA analysis, and two key gene modules were identified, containing 1 148 genes in total. Then, functional enrichment analysis was carried out for these genes in the modules. Venn diagrams, PPI network construction analysis, and candidate gene screening were employed. Finally, survival analysis was performed using TCGA and GTEx data in the GEPIAS database, and key genes associated with CRC were identified. The expression of BIGH3 gene was validated in colorectal cancer tissues by real-time fluorescence quantitative polymerase chain reaction (RTFQ-PCR), and its association with colorectal cancer was verified through clonogenic, scratch, and transwell assays in HCT116 and SW620 cell lines. Results: Through the analysis of CRC datasets from GEO, we screened a total of 2 214 differential genes. With the help of WGCNA analysis and PPI network construction, 4 CTC-related genes associated with CRC were identified. Survival analysis from the GEPIA database revealed that FCGBP and BIGH3 are associated with overall survival and disease-free survival. Further experiments indicated that BIGH3 gene is overexpressed in 30 matched colorectal cancer samples. The downregulation of BIGH3 expression could slow down cell proliferation and migration rates, as well as decrease invasiveness in HCT116 and SW620 cell lines. In contrast, upregulation of BIGH3 expression could increase its invasiveness or accelerate migration rate. Conclusion: FCGBP and BIGH3 are positively correlated with TNM staging, indicating their pivotal roles in CRC progression. They bear good prognostic value and may serve as potential biological markers for CRC clinically, and could provide potential therapeutic targets. Moreover, our experimental data revealed the crucial role of BIGH3 in colorectal cancer, suggesting it may influence the biological behavior of this disease.
[Keywords] Colorectal cancer; Circulating tumor cells; Biomarkers
结直肠癌(colorectal cancer,CRC)已成为全球恶性肿瘤中的第二大死因[1]。虽然肿瘤的综合治疗手段在近年取得了显著进展,但CRC患者的5年总生存情况仍然较差[2-3]。基因突变在肿瘤发生、发展中起到了关键作用,也是潜在的诊断标志物与治疗的靶点。例如,在CRC中REG4基因与侵袭性表型、肿瘤的恶性程度、淋巴结转移及耐药性形成具有相关性[4]。KRAS基因12号密码子的突变则是转移性CRC的独立预后因素[5]。也有研究发现,GLI3、ZEB1、PTPRB和TWIST1等基因可以调控细胞中的癌基因的转录并促进肿瘤发生、发展[6-8]。这些发现为探寻基于血液的CRC转移生物标志物提供了基础。然而,目前很多研究主要是通过比较肿瘤组织样本和正常组织样本来获得。考虑到CRC的复杂性和异质性,以及肿瘤本身的进展,循环肿瘤细胞(circulating tumor cell,CTC)检测技术为CRC的诊断和监测引入新的视角。CTC从原发肿瘤脱落并进入循环系统,甚至能在远处器官定植形成转移瘤[9]。多项研究证实CTC确实存在于CRC患者体内,且CTC具有作为CRC患者预后标志物的价值[10-12]。本研究通过生物信息学分析基因表达综合数据库(Gene Expression Omnibus,GEO)数据库中CTC数据集,筛选出与结直肠癌预后相关的基因FCGBP和BIGH3,可能作为潜在的预后标志物。通过结直肠癌组织验证表达以及肿瘤功能实验证实BIGH3与结直肠癌相关,可能是潜在的治疗靶点。
1 材料和方法
1.1 数据来源
从GEO数据库(http://www.ncbi.nlm.nih.gov/geo)中获取了数据集GSE74369、GSE117606和GSE164191。其中,GSE74369用于CTC基因表达分析,GSE117606用于WGCNA分析,GSE164191用于验证候选基因的表达。
1.2 差异表达基因分析
从GEO数据库下载基因表达矩阵和相应的注释文件。使用平台的注释信息将探针转换为同源基因符号。通过R语言“Limma”包[13]得到CRC患者邻近黏膜和肿瘤样本之间的DEG。截断标准为虚假发现率(false discovery rate,FDR)校正后P<0.01和差异表达倍数logFC>2。
1.3 WGCNA分析
使用R语言“WGCNA”包[14]识别CRC发病机制中的关键基因。首先,选择网络构建的软阈值,使共表达网络符合幂律分布并接近无标度网络。软阈值将邻接矩阵转换为0到1之间的连续值。其次,将邻接矩阵转换为拓扑重叠矩阵(topological overlap matrix,TOM)。使用TOM进行不相似性测量将基因划分为不同的基因模块。通过模块划分分析,使用动态树切割算法将基因按相似的表达模式进行聚类。不同基因模块以不同颜色进行标记实现可视化。然后测量每个基因模块的模块特征向量。确定与临床病理学特征最相关的基因模块。然后使用选定基因模块中的基因进行生物功能筛选。
1.4 功能富集分析
使用R语言 “clusterprofiler”包[15]进行GO富集分析和KEGG信号转导通路分析。
1.5 PPI网络构建和关键基因筛选
使用STRING 软件[16]生成蛋白质-蛋白质相互作用网络,探索相关的基因模块中基因之间的相互作用。这些基因符合交互作用得分≥0.4和最大互作数为0的条件。将基因导入Cytoscape软件(版本3.7.2)以构建可视化网络,并分析网络的拓扑特征。
1.6 生存分析
使用GEPIA数据库在线分析[17]评估关键基因对CRC患者的总生存期(overall survival,OS)和无病生存期(disease-free survival,DFS)的影响,在分析中使用了癌症基因组图谱(The Cancer Genome Atlas,TCGA)和基因型-组织表达数据库(Genotype-Tissue Expression,GTEx)中的肿瘤组织样本和正常组织样本的转录组测序数据。
1.7 组织收集
本研究包括了2022年1月—2023年12月在镇海区人民医院接受治疗的30例结直肠癌患者。患者在手术前均未接受化疗或放疗。手术后,即刻将切除的结直肠癌组织和匹配的邻近组织样本在液氮中冷冻,然后转移到-80 ℃冰箱中稳定储存直至使用。所有患者都签署了知情同意书。
1.8 试剂
DMEM、L-15培养基购自美国Gibco公司,胎牛血清(fetal bovine serum,FBS)购自德国PAN-Biotech公司,1%的青霉素/链霉素购自美国Hyclone公司,LipofectamineTM2000、TRIzol试剂购自美国Invitrogen公司,反转录试剂盒及实时荧光定量聚合酶链反应(real-time fluorescence quantitative polymerase chain reaction,RTFQ-PCR)预混液购自苏州近岸蛋白质科技股份有限公司,GAPDH引物购自北京擎科生物科技有限公司,4%多聚甲醛购自北京兰杰柯科技有限公司,结晶紫溶液购自北京索莱宝科技有限公司,移液器吸头购自美国Axygen公司,transwell小室购自美国Corning公司。
1.9 细胞系培养与转染
人结直肠癌细胞系HCT116细胞用DMEM培养基培养,人结直肠癌细胞系SW620细胞用L-15培养基培养。所有培养基补充有10%的胎牛血清和1%的青霉素/链霉素。细胞在37 ℃、CO2体积分数为5%的培养箱中培养。根据说明书使用LipofectamineTM2000转染试剂将PCD5-BIGH3、si-BIGH3转染到细胞中,转染48 h后用于后续实验。
1.10 RNA制备与RTFQ-PCR检测
按照说明书步骤使用TRIzol试剂从组织或细胞系中提取总RNA。使用反转录试剂盒将微小RNA(microRNA)反转录成cDNA,再对cDNA进行扩增,RTFQ-PCR引物序列见表1,GAPDH被用作内参。小干扰RNA序列见表2。
1.11 克隆形成实验
将转染后的细胞以每孔1 000个细胞密度接种在6孔板中。每隔5天换培养基,培养2周后,将细胞用4%多聚甲醛固定15 min,然后用1%的结晶紫溶液染色15 min,最后统计细胞个数。
1.12 划痕实验
将各组转染后的细胞在6孔板中培养至90%融合度时,使用200 μL移液器吸头在无菌直尺的帮助下划出相同宽度的划痕,使用显微镜拍摄图像,此时记为0 h。然后将细胞在无血清培养基中温育48 h并再次拍照,记录每个时间点的伤口宽度。用ImageJ软件评估细胞迁移率。
1.13 Transwell迁移实验
将各组计数好的细胞用200 μL无血清培养基重悬后置于transwell上室,并在下室中加入750 μL含10%的FBS培养基。温育48 h后,使用棉签去除上室内的细胞。侵入膜底部的细胞用4%的多聚甲醛固定15 min,再用1%的结晶紫染色。使用显微镜拍照,然后使用ImageJ软件评估细胞数量。
2 结 果
2.1 CTC与正常对照的DEG鉴定及功能富集分析
在GSE74369数据集中,将18个CTC样本和15个正常组织样本进行差异分析,使用FDR<0.01和logFC>2的截断标准,共筛选出了2 214个DEG(图1A,B)。对DEG进行功能考证,GO富集分析和KEGG信号转导通路分析显示与肿瘤发展密切相关,其中大部分与粘着斑、细胞外基质及脂肪酸代谢通路有关(图1C、D)。
图1 基于GEO数据库对CTC的差异表达基因进行分析
Fig. 1 Differently expressed genes of CTC were analyzed based on GEO database
2.2 WGCNA网络构建及关键基因模块识别
使用GSE117606数据集中RNA-seq表达数据构建WGCNA网络,将软阈值设为5,使构建的共表达网络接近无标度状态。WGCNA分析确定了13个共表达关键基因模块。这13个关键基因模块与CRC的原发病灶(T)、淋巴结转移(N)以及远处转移(M)等临床特征相关。我们发现黑色模块中的基因与CRC原发灶临床特征显著相关(R=0.76,P<0.001)。此外,基于WGCNA的特征向量相邻度矩阵显示了不同模块之间的相关性。通过各个模块特征向量与临床特征之间的相关分析得到相关性。
本研究基于DEGs和WGCNA分析共获得180个CTC中与进展高度相关的基因,进一步构建了PPI网络。为了缩小我们的研究范围,我们筛选了Degree≥4的基因,在网络中保留了53个基因。使用TCGA数据库,发现25个基因在CRC肿瘤与正常样本中表达差异有统计学意义。生存分析表明,FCGBP、BIGH3、IL1B和NAT1可能作为潜在的CRC生物标志物。
2.3 FCGBP和BIGH3基因与CRC患者的生存相关
本研究使用GEPIA数据库评估了4个关键基因,其中FCGBP和BIGH3的表达与CRC肿瘤分期具有相关性。结果表明,FCGBP的表达随着肿瘤分期的升高呈显著降低趋势(P<0.05),而BIGH3的表达则随着肿瘤分期增加而升高(图2A、B),表明FCGBP和BIGH3与CRC患者的肿瘤进展具有相关性。此外,本研究进一步探索了FCGBP和BIGH3在CRC患者中的预后预测价值。使用Kaplan-Meier曲线和log-rank检验进行的生存分析进一步发现FCGBP和BIGH3的mRNA表达水平与生存率相关。其中,FCGBP高表达组患者的OS和DFS均高于低表达组(图2C、D)。BIGH3高表达组的DFS显著高于低表达组(图2E),然而BIGH3表达水平在OS中差异无统计学意义(图2F)。
图2 基于GEPIA进行FCGBP、BIGH3对结直肠癌患者的肿瘤分期和生存分析的影响
Fig. 2 Tumor stages and survival analysis of FCGBP and BIGH3 for CRC patients were performed based on GEPIA
2.4 BIGH3基因在HCT116和SW620细胞系中促进增殖、迁移与侵袭
采用RTFQ-PCR在30对结直肠癌与癌旁配对样本中检测了FCGBP和BIGH3基因表达发现, BIGH3基因在结直肠癌组织中的表达相比于癌旁组织升高(P<0.001)。针对BIGH3基因设计了si-RNA与过表达质粒,并通过RTFQ-PCR在HCT116和SW620细胞系中进行敲低与过表达验证(图3A)。本研究选用了敲低效果较好的si-BIGH3#2小干扰RNA以及过表达质粒进一步对HCT116和SW620细胞系进行基因敲低与上调,并通过克隆形成、划痕实验以及transwell实验等功能实验进一步探究BIGH3基因对结直肠癌细胞生物学行为的影响。结果显示,在HCT116细胞系中下调BIGH3基因表达后,可使克隆形成实验中细胞增殖速度减少以及划痕实验中细胞迁移率降低,而上调BIGH3基因表达后在transwell实验中表现出更强的侵袭性(图3B)。在SW620细胞系中下调BIGH3基因表达后,可使克隆形成实验中细胞增殖速度减少且在transwell实验中的侵袭性减弱,上调BIGH3基因表达后在划痕实验中表现出更高的细胞迁移率(图3C)。
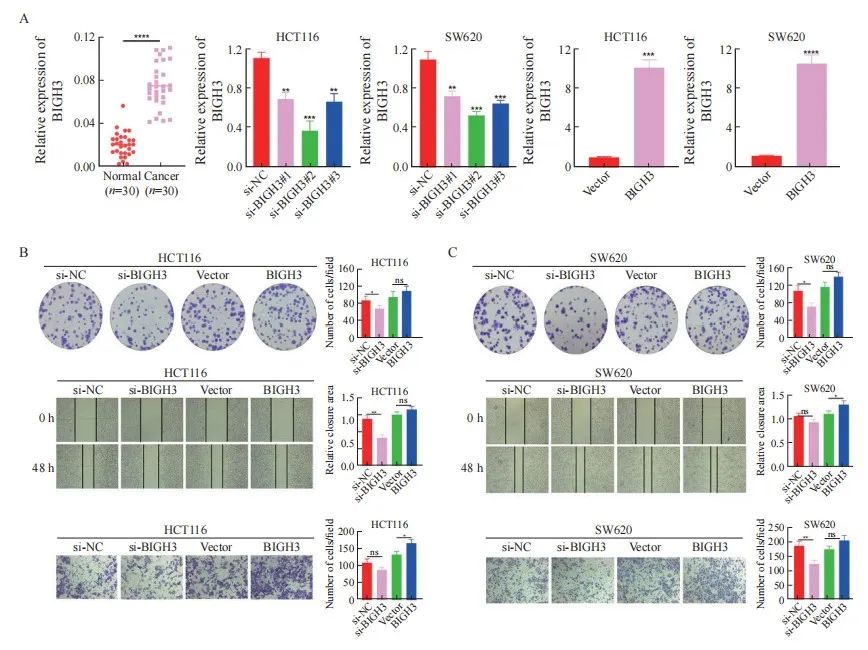
图3 BIGH3基因在HCT116和SW620细胞系中促进增殖、迁移与侵袭
Fig. 3 The BIGH3 gene promotes proliferation, migration, and invasion in HCT116 and SW620 cell lines
3 讨 论
CTC由肿瘤脱落入血并能在远处器官定植,其可在血液中被检测发现[18]。一项研究分析了前列腺癌的CTC转录本测序结果,发现可以预测转移[19]。在胰腺癌患者的CTC中,高表达的CLIC4和GAS2L1可能作为判定肿瘤进展的生物标志物[20]。这些在CTC中发现的特征基因可以预测肿瘤的进展,并指导临床治疗,同时也可以作为预测患者预后的潜在生物标志物。
为了从CTC的角度探索CRC进展过程中的关键基因,我们首先使用与CTC相关的数据集进行探索。结果显示,在CTC和正常样本之间存在2 155个DEG。富集分析显示,这些DEG主要与钙黏附、生长因子结合、碳代谢、化学致癌物质、脂肪酸相关通路及细胞外基质相互作用有关。研究表明,代谢异常是肿瘤的新型生物标志物,特别是脂肪酸代谢,其与CRC的发生、发展密切相关[21-24]。基于GSE117606数据集筛选出482个CRC相关DEG,这些基因在结直肠肿瘤和正常对照样本之间具有显著差异,用于后续的WGCNA分析。在我们的研究中,WGCNA结果中黑色和蓝色模块共包含1 148个基因,被确定为与CRC的临床特征最为相关。此外,PPI网络、TCGA表达和生存分析均证明了FCGBP和BIGH3是与CRC发展密切相关的两个关键基因(图4)。通过进一步实验验证,BIGH3基因在结直肠癌细胞系中起着关键作用。BIGH3在30对结直肠癌与癌旁配对样本的检测中表现出高表达,提示其可能对于癌症发展有促进作用。我们在HCT116和SW620两个结直肠癌细胞系的进一步的功能实验也证实了这一猜测,敲低BIGH3的表达能够减慢细胞增殖和迁移的速度,并降低侵袭性。相反,上调BIGH3的表达会增加其侵袭性或提高迁移率。BIGH3基因是转化生长因子β(transforming growth factor-β,TGF-β)的下游靶基因,与多种肿瘤的发生、发展高度相关[25-27]。在CRC的肿瘤细胞中,TGF-β诱导BIGH3促进血管生成,从而起到促进CRC转移的作用[28]。有研究[29]也显示BIGH3高表达是结直肠癌转移的关键因素,这与本研究结果一致。
利用CTC数据集和WGCNA分析,本研究确定了与CRC高度相关的基因模块。生物信息学分析证明,FCGBP和BIGH3与CRC发生、发展密切相关,并与肿瘤分期呈正相关,具有较好的预后预测价值。本研究的实验数据也揭示了BIGH3在结直肠癌中的重要角色,它可能影响结直肠癌的生物学行为,并可能作为潜在的治疗靶点。
利益冲突声明:所有作者均声明不存在利益冲突。
作者贡献声明:葛祖荫负责实验设计、数据分析、文章撰写;宋坤负责样本收集与整理、数据分析、文章撰写;林云霄负责样本收集、临床资料管理;钟烨凌负责数据收集与整理、统计,以及实验操作;郝敬铎负责研究设计、论文审阅、修改、经费支持。
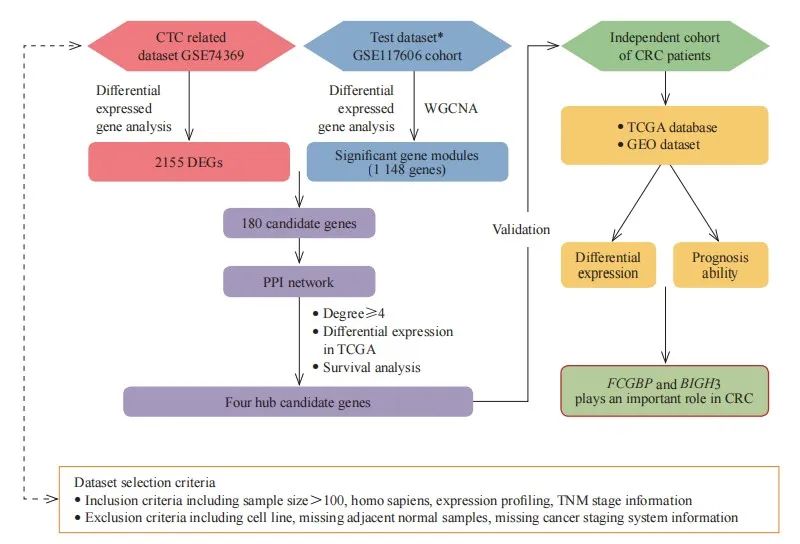
图4 本文CTC分析流程图
Fig. 4 Flow chart of CTCs in this study
[参考文献]
[1] BRAY F, LAVERSANNE M, SUNG H, et al. Global cancer statistics 2022: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2024, 74(3): 229-263.
[2] MASUISHI T, TANIGUCHI H, HAMAUCHI S, et al. Regorafenib versus trifluridine/tipiracil for refractory metastatic colorectal cancer: a retrospective comparison[J]. Clin Colorectal Cancer, 2017, 16(2): e15-e22.
[3] ZHOU L, WANG J Z, WANG J T, et al. Correlation analysis of MR/CT on colorectal cancer lymph node metastasis characteristics and prognosis[J]. Eur Rev Med Pharmacol Sci, 2017, 21(6): 1219-1225.
[4] ZHANG J Y, ZHU Z, MIAO Z F, et al. The clinical significance and mechanisms of REG4 in human cancers[J]. Front Oncol, 2020, 10: 559230.
[5] DOLATKHAH R, DASTGIRI S, EFTEKHAR SADAT A T, et al. Impact of RAS/RAF mutations on clinical and prognostic outcomes in metastatic colorectal cancer[J]. Bioimpacts, 2021, 11(1): 5-14.
[6] SHEN M Y, ZHANG Z Y, WANG P. GLI3 promotes invasion and predicts poor prognosis in colorectal cancer[J]. Biomed Res Int, 2021, 2021: 8889986.
[7] YE D X, WANG S S, HUANG Y, et al. USP43 directly regulates ZEB1 protein, mediating proliferation and metastasis of colorectal cancer[J]. J Cancer, 2021, 12(2): 404-416.
[8] WENG X Y, CHEN W, HU W X, et al. PTPRB promotes metastasis of colorectal carcinoma via inducing epithelialmesenchymal transition[J]. Cell Death Dis, 2019, 10(5): 352.
[9] FERREIRA M M, RAMANI V C, JEFFREY S S. Circulating tumor cell technologies[J]. Mol Oncol, 2016, 10(3): 374-394.
[10] BORK U, RAHBARI N N, SCHÖLCH S, et al. Circulating tumour cells and outcome in non-metastatic colorectal cancer: a prospective study[J]. Br J Cancer, 2015, 112(8): 1306-1313.
[11] COHEN S J, PUNT C J A, IANNOTTI N, et al. Relationship of circulating tumor cells to tumor response, progressionfree survival, and overall survival in patients with metastatic colorectal cancer[J]. J Clin Oncol, 2008, 26(19): 3213-3221.
[12] HUANG X Z, GAO P, SONG Y X, et al. Relationship between circulating tumor cells and tumor response in colorectal cancer patients treated with chemotherapy: a meta-analysis[J]. BMC Cancer, 2014, 14: 976.
[13] RITCHIE M E, PHIPSON B, WU D, et al. Limma powers differential expression analyses for RNA-sequencing and microarray studies[J]. Nucleic Acids Res, 2015, 43(7): e47.
[14] LANGFELDER P, HORVATH S. WGCNA: an R package for weighted correlation network analysis[J]. BMC Bioinformatics, 2008, 9: 559.
[15] YU G C, WANG L G, HAN Y Y, et al. clusterProfiler: an R package for comparing biological themes among gene clusters[J]. OMICS, 2012, 16(5): 284-287.
[16] SZKLARCZYK D, GABLE A L, LYON D, et al. STRING v11: protein-protein association networks with increased coverage, supporting functional discovery in genome-wide experimental datasets[J]. Nucleic Acids Res, 2019, 47(D1): D607-D613.
[17] TANG Z F, LI C W, KANG B X, et al. GEPIA: a web server for cancer and normal gene expression profiling and interactive analyses[J]. Nucleic Acids Res, 2017, 45(W1): W98-W102.
[18] QUIROZ-REYES A G, ISLAS J F, DELGADO-GONZALEZ P, et al. Therapeutic approaches for metastases from colorectal cancer and pancreatic ductal carcinoma[J]. Pharmaceutics, 2021, 13(1): 103.
[19] MIYAMOTO D T, LEE R J, KALINICH M, et al. An RNAbased digital circulating tumor cell signature is predictive of drug response and early dissemination in prostate cancer[J]. Cancer Discov, 2018, 8(3): 288-303.
[20] ZHU L, HISSA B, GYŐRFFY B, et al. Characterization of stem-like circulating tumor cells in pancreatic cancer[J]. Diagnostics, 2020, 10(5): 305.
[21] PENG Y R, XU C X, WEN J, et al. Fatty acid metabolismrelated lncRNAs are potential biomarkers for predicting the overall survival of patients with colorectal cancer[J]. Front Oncol, 2021, 11: 704038.
[22] GILKES D M, SEMENZA G L, WIRTZ D. Hypoxia and the extracellular matrix: drivers of tumour metastasis[J]. Nat Rev Cancer, 2014, 14(6): 430-439.
[23] HANLEY C J, NOBLE F, WARD M, et al. A subset of myofibroblastic cancer-associated fibroblasts regulate collagen fiber elongation, which is prognostic in multiple cancers[J]. Oncotarget, 2016, 7(5): 6159-6174.
[24] RĂCHIERIU C, ENIU D T, MOIŞ E, et al. Lipidomic signatures for colorectal cancer diagnosis and progression using UPLCQTOF-ESI+MS[J]. Biomolecules, 2021, 11(3): 417.
[25] CALAF G M, ECHIBURÚ-CHAU C, ZHAO Y L, et al. BigH3 protein expression as a marker for breast cancer[J]. Int J Mol Med, 2008, 21(5): 561-568.
[26] ZHAO Y L, EL-GABRY M, HEI T K. Loss of Betaig-h3 protein is frequent in primary lung carcinoma and related to tumorigenic phenotype in lung cancer cells[J]. Mol Carcinog, 2006, 45(2): 84-92.
[27] PAN T H, LIN S C, YU K J, et al. BIGH3 promotes osteolytic lesions in renal cell carcinoma bone metastasis by inhibiting osteoblast differentiation[J]. Neoplasia, 2018, 20(1): 32-43.
[28] CHIAVARINA B, COSTANZA B, RONCA R, et al. Metastatic colorectal cancer cells maintain the TGFβ program and use TGFBI to fuel angiogenesis[J]. Theranostics, 2021, 11(4): 1626-1640.
[29] PAN X, MA X X. A novel six-gene signature for prognosis prediction in ovarian cancer[J]. Front Genet, 2020, 11: 1006.
作者:中国癌症杂志
版权声明:
本网站所有注明“来源:梅斯医学”或“来源:MedSci原创”的文字、图片和音视频资料,版权均属于梅斯医学所有。非经授权,任何媒体、网站或个人不得转载,授权转载时须注明“来源:梅斯医学”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
在此留言










#结直肠癌# #循环肿瘤细胞# #生物标志物#
43